Class 12 Biology Chapter 6 वंशागति का आणविक आधार in Hindi Notes: कक्षा 12 जीव विज्ञान अध्याय 6: वंशानुक्रम का आणविक आधार के लिए व्यापक हिंदी नोट्स एक्सेस करें। सरलीकृत स्पष्टीकरण और प्रमुख अवधारणाओं को शामिल किया गया।
Class 12 Biology Chapter 6 वंशागति का आणविक आधार in Hindi Notes
डीएनए
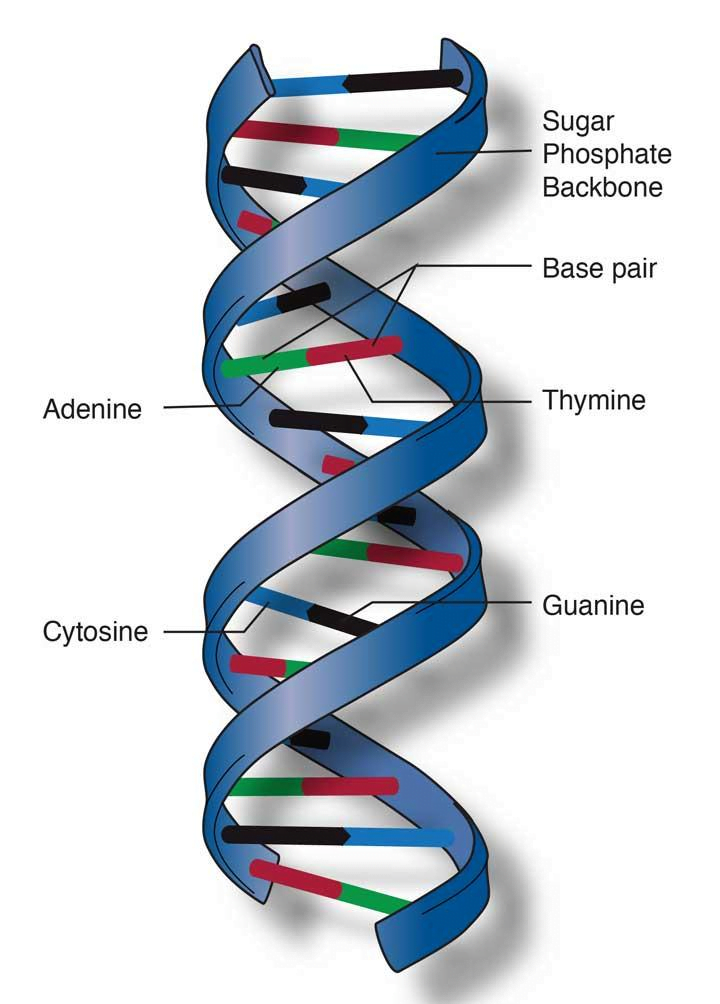
डीएनए की संरचना का परिचय (Introduction):-
कोशिका में केंद्रक (Nucleus) होता है इस केन्द्रक में गुणसूत्र Choromosome पाये जाते हैं, जिनमें जीन होते हैं।
जीन आनुवंशिक सूचना के वाहक होते हैं। जीन स्वयं DNA का छोटा खण्ड है।
जीवाणुभोजी (Bacteriophage) 174 में 5386 न्यूक्लियोटाइड पाए जाते हैं।
जीवाणुभोजी लैब्डा में 48502 क्षार युग्म (base pair) जीवाणु इस्चेरिचया कोलाई में 4.6×106 क्षार युग्म व मनुष्य के अगुणित डीएनए में 3.3×109 क्षार युग्म होते है।
डीएनए की रासायनिक प्रकृति (The Chemical Nature of DNA)
डीएनए का पूरा नाम डिऑक्सीराइबोज न्यूक्लिक अम्ल (Deoxyribose Nucleic Acid) है। यह आनुवंशिक पदार्थ है। जो सभी जीवों की कोशिकाओं में पाया जाता है। प्रोकेरियोट में यह वृताकार (Circular) जबकि यूकेरियोट में यह रैखिक (Linear) होता है।

DNA एक बहुलक (Polymer) है। जो पॉलीन्यूक्लियोटाइड की दो श्रृंखलाओं का बना होता है। पॉलीन्यूक्लियोटाइड श्रृंखलाएं न्यूक्लियोटाइड इकाइयों से मिलकर बनी होती है। प्रत्येक न्यूक्लियोटाइड की तीन उप इकाइयाँ होती है।
पेण्टॉज शर्करा (5 कार्बन शर्करा) (Pantose Sugar)-
यह 5C युक्त कार्बोहायड्रेट है। इसे डिऑक्सीराइबोज शर्करा भी कहते हैं, क्योंकि राइबोज शर्करा के दूसरे कार्बन पर -OH (हाइड्रोक्सिल) की जगह पर केवल -H (हाइड्रोजन) पाया जाता है। इसको β-2′ डिऑक्सीराइबोज शर्करा भी कह सकते है।
नाइट्रोजन क्षार (Nitrogen Base) –
ये दो प्रकार के होते है-
- प्यूरीन क्षार (Purine):-

ये नाइट्रोजन युक्त दो वलय से बनी होती है। जिनमें एक हेक्सा वलय जिसे पाइरिमिडीन वलय तथा दूसरी वलय पेण्टा वलय होती है। जिसे इमीडीजोल वलय कहते है। ऐडेनीन (A) तथा ग्वानीन (G) प्यूरीन क्षार के अंतर्गत आते है।
B) पाइरिमिडीन क्षार (Pyrimidine): –
इनमें केवल एक वलय हेक्सा वलय (पाइरिमिडीन वलय ) पायी जाती है। इसमें थाइमीन (T), साइटोसीन (C), तथा युरेसिल (U) सम्मिलित है।

पेण्टॉज शर्करा तथा नाइट्रोजन क्षार ग्लाइकोसिडिक बंध (glycosidic bond) द्वारा जुड़कर न्यूक्लियोसाइड का निर्माण करते हैं।
पेण्टॉज शर्करा + नाइट्रोजन क्षार = न्यूक्लियोसाइड
उदाहरण-
- एडिनिन + डिऑक्सीराइबोज शर्करा = डिऑक्सीएडिनोसिन (DNA)
- ग्वानिन + डिऑक्सीराइबोज शर्करा = डिऑक्सीग्वानोसिन (DNA)
- साईटोसिन + डिऑक्सीराइबोज शर्करा = डिऑक्सीसाईटीडीन (DNA)
- थायमिन + डिऑक्सीराइबोज शर्करा = डिऑक्सीथायमिडीन (DNA)
C) फॉस्फेट समूह (PO4 ) (Phosphate Group)-
यह H3PO4 से प्राप्त होता है। न्यूक्लियोसाइड फॉस्फोरिक अम्ल से फॉस्फोएस्टर बंध से जुड़कर कर न्यूक्लिओटाइड बनते है।
पेण्टॉज शर्करा + नाइट्रोजन क्षार + फॉस्फेट समूह = न्यूक्लिओटाइड
उदाहरण –
- डिऑक्सीएडिनिलिक अम्ल or Deoxyadenylate
- डिऑक्सीग्वानिलिक अम्ल or Deoxythymidylate
- डिऑक्सीसाईटीडीलिक अम्ल or Deoxycytidylate
- डिऑक्सीथायमिडीलिक अम्ल or Deoxyguanylate
पॉलीन्यूक्लियोटाइड (Polynucleotide): –
दो न्यूक्लियोटाइड आपस में 3’-5’ फॉस्फोडाईएस्टर बंध से जुड़कर डाईन्यूक्लियोटाइड का निर्माण करते है, और कई न्यूक्लियोटाइड आपस में फॉस्फोडाईएस्टर बंध द्वारा जुड़कर पॉलीन्यूक्लियोटाइड का निर्माण करते है। दो पॉलीन्यूक्लियोटाइड श्रृंखलाओं के प्युरिन तथा पाइरिमिडीन एक दूसरे से हाइड्रोजन बंध से जुड़कर कुंडलित होकर डीएनए बनाते है।

एडिनिन, थायमिन से दो हाइड्रोजन बंध के द्वारा तथा ग्वानिन, साईटोसिन से तीन हाइड्रोजन बंध के द्वारा जुड़ा होता है।
DNA की भौतिक संरचना (The Physical Structure of DNA)–
डीएनए अणु त्रिविमीय होता है। और दो रज्जुक (Strain)) ओं से बना होता है। जो कि एक दूसरे के चारों ओर कुंडलित होते हैं। आल्टमान ने इनको न्यूक्लिक अम्ल नाम दिया फ्रैंकलिन और विल्किन्स ने DNA के X किरण विवर्तन के अध्ययन से यह दर्शाया है। कि DNA द्विकुंडिलत होता है। 1953 में जेम्स वाटसन व फैंसिस क्रिक को DNA की संरचना की खोज करने के लिये नोबेल पुरस्कार दिया गया। वाटसन और क्रिक मॉडल के अनुसार-

- DNA अणु दो कुंडलियों (DOUBLE HELIX ) से निर्मित हैं जिसमें DNA के दो रज्जुक (Strain) होते हैं। दोनों रज्जुक (Strain) प्रतिसमांतर रूप में रहते हैं जिसका आशय यह हुआ कि एक रज्जुक (Strain) में न्यूक्लियोटाइड का अनुक्रम 5’ से 3’ की दिशा में और दूसरे रज्जुक (Strain)) में 3’ से 5’ की दिशा में होता है। (3’ व 5’ का आशय उन कार्बन परमाणुओं से है। जिससे फॉस्फेट समूह जुड़े रहते हैं।)
- कुण्डली का आधार शर्करा फॉस्फेट से निर्मित होता है। और नाइट्रोजन क्षार शर्करा से सहलग्न होते हैं।
- दोनों रज्जुकों (Strain) के क्षार हाइड्रोजन बंधों द्वारा जुड़े होते हैं।
4. A व T के बीच दो हाइड्रोजन बंध तथा G व C के बीच तीन हाइड्रोजन बंध होते हैं। एक DNA कुण्डली में एक पूरा कुण्डलीय घुमाव 3.4Nm (नैनोमीटर) या 34 Å के लम्बा होता है। इस पूरे घुमाव में 10 क्षार युग्म होते हैं। प्रत्येक क्षार युग्म परस्पर 0.34 nm (3.4Å) दूरी से पृथक्कृत होते हैं। दोहरे कुण्डलित DNA अणु का व्यास 2nm होता है। वाटसन व क्रिक मॉडल इस बात की भलीभाँति व्याख्या करता है। कि किस प्रकार अणु DNA के दो रज्जुक (Strain), प्रतिकृति (REPLICATION) व अनुलेखन (TRANSCRIPTION) के दौरान पृथक होकर पुनः कुंडलित हो सकते हैं।
चारगाफ का नियम
चारगाफ के नियमानुसार क्षार युग्मन अति विशिष्ट होता है। एक एडेनीन (ADENINE) प्यूरीन क्षार सदैव थाइमीन (THYMINE) – पिरिमिडीन क्षार के साथ युग्मित होता है। प्यूरीन क्षार ग्वानीन (GUANINE) – पिरिमिडीन क्षार, साइटोसीन (CYTOSINE) के साथ संयुक्त होता है। क्षार के ये युग्म पूरक क्षार (COMPLEMENTARY BASE) कहलाते हैं।
डीएनए के प्रकार (Type of DNA) –
जीवों में मुख्यतः तीन प्रकार के DNA पाये जाते है –

A-DNA –
इस प्रकार के DNA का कुंडलित रज्जुक छोटा, चौड़ा, तथा एक गहरे बड़े व छोटे उथले खांच युक्त होता है। इसमें दोनों खांचो की गहराई समान होती है। A-DNA वामावर्त कुंडलित होता है। इसमें प्रत्येक कुंडलन में 10.9 / 11 क्षार युग्म पाये जाते है। तथा क्षार, कुंडलन की मुख्य अक्ष से 13० दूरी पर होते है।
B-DNA –
इस प्रकार के DNA का कुंडलित रज्जुक पतला व लम्बा होता है। इसमें एक खांच गहरी तथा दूसरी उथली होती है। B-DNA वामावर्त कुंडलित होता है। इसमें प्रत्येक कुंडलन में 10.9 / 11 क्षार युग्म पाये जाते है। तथा क्षार, कुंडलन की मुख्य अक्ष से 2० दूरी पर होते है।
Z-DNA –
इस प्रकार के DNA का कुंडलित रज्जुक पतला, लम्बा, तथा एक गहरी खांच युक्त होता है। Z-DNA दक्षिणावर्त कुंडलित होता है। इसमें प्रत्येक कुंडलन में 12 क्षार युग्म पाये जाते है। यह टेढ़ा-मेढ़ा दिखाई देने के कारण इसे Z-DNA कहते है|
डीएनए की रासायनिक प्रकृति
DNA एक बहुलक है जो पॉलीन्यूक्लियोटाइड की दो श्रंखलाओ का बना होता है है पॉलीन्यूक्लियोटाइड श्रंखलाओ न्यूक्लियोटाइड इकाइयों से मिलकर बनी है। प्रत्येक न्यूक्लियोटाइड की तीन उप इकाइयाँ होती है।
1. पेन्टोस शर्करा (5 कार्बन शर्करा)-
यह 5C युक्त कार्बोहायड्रेट है इसे डिऑक्सीराइबोज शर्करा भी कहते हैं। क्योंकी राइबोज शर्करा के दुसरे कार्बन पर -OH (हाइड्रोक्सिल) की जगह पर केवल -H (हाइड्रोजन) पाया जाता है। इसको 2′ डिऑक्सीराइबोज शर्करा भी कह सकते है
2. नाइट्रोजनी क्षारक (क्षार)-
ये दो प्रकार के होते है
A) प्यूरीन क्षार
ये नाइट्रोजन युक्त दो वलय से बनी होती है जिनमें एक हेक्सा वलय जिसे पाइरिमिडीन वलय तथा दूसरी वलय पेण्टा वलय होती है जिसे इमीडीजोल वलय कहते है ऐडेनीन (A) तथा ग्वानीन (G) प्यूरीन क्षार के अंतर्गत आते है
B) पाइरिमिडीन क्षार
इनमें केवल एक वलय हेक्सा वलय (पाइरिमिडीन वलय ) पायी जाती है इसमें थाइमीन (T), साइटोसीन (C), तथा युरेसिल (U) सम्मिलित है।
फॉस्फेट समूह (PO4 )- जो H3PO4 से प्राप्त होता है।
एक क्षार और शर्करा मिलकर एक न्यूक्लियोसाइड का निर्माण करते हैं, एक फॉस्फेट समूह के इसके साथ संयुक्त हो जाने से यह एक न्यूक्लिओटाइड बन जाता है।
DNA की भौतिक संरचना-
DNA अणु त्रिविमीय होता है और दो तंतुओं से बना होता है जोकि एक दूसरे के चारों ओर कुंडलित होते हैं। फ्रैंकलिन और विल्किन्स ने DNA के X किरण विवर्तन के अध्ययन से यह दर्शाया है कि DNA द्विकुंडिलत होता है। 1953 में जेम्स वाटसन व फैंसिस क्रिक को DNA की संरचना की खोज करने के लिये नोबेल पुरस्कार दिया गया। वाटसन और क्रिक मॉडल के अनुसार-
- DNA अणु दो कुंडलियों (DOUBLE HELIX ) से निर्मित हैं जिसमें DNA के दो तंतु होते हैं। दोनों तंतु प्रतिसमांतर रूप में रहते हैं जिसका आशय यह हुआ कि एक तंतु में न्यूक्लियोटाइड का अनुक्रम 5’ से 3’ की दिशा में और दूसरे तंतु में 3’ से 5’ की दिशा में होता है। (3’ व 5’ का आशय उन कार्बन परमाणुओं से है जिससे फॉस्फेट समूह जुड़े रहते हैं।)
- कुण्डली का आधार शर्करा फॉस्फेट से निर्मित होता है और नाइट्रोजनी क्षारक शर्करा से सहलग्न होते हैं।
- दोनों तंतुओं के क्षारक हाइड्रोजन बंधों द्वारा जुड़े होते हैं।
- शार्गपफ़ के नियमानुसार क्षारक युग्मन अति विशिष्ट होता है। एक एडेनीन (ADENINE) प्यूरीन क्षारक सदैव थाइमीन (THYMINE) – पिरिमिडीन क्षारक के साथ युग्मित होता है। प्यूरीन क्षारक ग्वानीन (GUANINE) – पिरिमिडीन क्षारक, साइटोसीन (CYTOSINE) के साथ संयुक्त होता है। क्षारक के ये युग्म पूरक क्षारक (COMPLEMENTARY BASE) कहलाते हैं।
- A व T के बीच दो हाइड्रोजन बंध तथा G व C के बीच तीन हाइड्रोजन बंध होते हैं। एक DNA कुण्डली में एक पूरा कुण्डलीय घुमाव 3.4Nm (नैनोमीटर) या 34 Å के लम्बा होता है। इस पूरे घुमाव में 10 क्षारक युग्म होते हैं। प्रत्येक क्षारक युग्म परस्पर 0.34 nm (3.4Å) दूरी से पृथक्कृत होते हैं। दोहरे कुण्डलित DNA अणु का व्यास 2nm होता है। वाटसन व क्रिक मॉडल इस बात की भलीभाँति व्याख्या करता है कि किस प्रकार अणु DNA के दो तंतु, प्रतिकृति (REPLICATION) व अनुलेखन (TRANSCRIPTION) के दौरान पृथक होकर पुनः कुंडलित हो सकते हैं।
डी एन ए की प्रतिकृति
- 5 ‘-> 3’ दिशा में DNA प्रतिकृति होती है।
- प्रत्येक न्यूक्लियोटाइड में 5 कार्बन युक्त एक डीऑक्सीरीइबॉज अणु होता है, कार्बन 1 से नाइट्रोजन बेस और कार्बन 5 से फॉस्फेट समूह जुड़ा
- डीएनए प्रतिकृति में केवल 3′ सिरा में डीएनए न्यूक्लियोटाइड जोड़ा जाता है
प्रोकेरीओट्स में DNA प्रतिकृति की प्रक्रिया
एंजाइम–
- Helicase: Coiled DNA को unwinding करती है DNA के पूरक रज्जुक को अलग करके एक प्रतिकृति द्विशाखा का निर्माण करती है

- RNA Primase: DNA Polymerase-III DNA न्यूक्लियोटाइड को केवल DNA के स्वतंत्र 3’ सिरे पर जोड़ने में सक्षम है इसलिए RNA Primase DNA टेम्पलेट का उपयोग एक छोटे 10 न्यूक्लियोटाइड अनुक्रम युक्त आरएनए को संश्लेषित करता है जो आरएनए प्राइमर के रूप में जाना जाता है
- DNA polymerase-III: डीएनए पोलीमरेज़-III पुर्ववर्ती डीएनए के एकल रज्जुक का उपयोग टेम्पलेट के रूप में करता है यह Dioxyribonucleoside triphosphate (dNTP) को DNA के टेम्पलेट रज्जुक पर पूरक आधार युग्मन नियमों (Complementary)के अनुसार (A=T, G=C) जोड़ता है डीएनए पोलीमरेज़- III डीएनए न्यूक्लियोटाइड को केवल DNA के स्वतंत्र 3’ सिरे पर को जोड़ सकता है DNA के दो रज्जुक में से केवल एक पर ही DNA लगातार संश्लेषित होता है: यह अग्रगामी रज्जुक के रूप में जाना जाता है
- DNA polymerase-I: DNA पोलीमरेज़ I एक प्रूफ़रीडिंग एंजाइम है यह आरएनए प्राइमर के आरएनए न्यूक्लियोटाइड को हटा देता है और उनकी जगह डीएनए न्यूक्लियोटाइड को जोड़ता है
- DNA ligase: ओकाज़ाकी खण्ड को सहसंयोजक बंधन के द्वारा जोड़ता है पश्चगामी रज्जुक (Lagging Strain) के ओकाज़ाकी खण्ड को जोड़कर DNA संश्लेषण पूरा करता है
- Topo-isomerase: यह DNA unwinding के समय अतिकुण्डलन को हटाता है और DNA में तनाव को कम करता है
- SSB protein(Single Strain Binding): यह खुले हूए DNA रज्जुक को पुनः जुङने से रोकता है
Okazaki Fragments
डीएनए पोलीमरेज़ III सीधे पश्चगामी रज्जुक (Lagging Strain) पर काम करने में असमर्थ है क्योंकि इसमें मौजूदा DNA किनारा पर एक मुक्त 3′ सिरा नहीं है इसलिए, संश्लेषण की शुरुआत तब शुरू होती है जब आरएनए प्राइमेज़ DNA टेम्पलेट का इस्तेमाल करके एक आरएनए प्राइमर (छोटे 10 आरएनए न्यूक्लियोटाइड अनुक्रम) को संश्लेषित करता है

DNA पोलीमरेज़ III तब आरएनए प्राइमर के मुक्त 3′ सिरा का उपयोग करता है ताकि DNA के अनुक्रमों को संश्लेषित किया जा सके (लगभग 100 डीएनए न्यूक्लियोटाइड्स) जिसे ओकाजाकी टुकड़े के रूप में जाना जाता है
Deoxyribonuleoside Triphosphate
फॉस्फेट समूह के हाइड्रोलिसिस के द्वारा न्यूक्लियोटाइड के बीच फोस्फोडाइस्टर सहसंयोजक बंधन बनाने के लिए ऊर्जा प्राप्त होती है

डीएनए अनुलेखन की प्रकिया
DNA से आनुवंशिक सुचनाओ का RNA में स्थानान्तरण अनुलेखन(transcription) कहलाता है।
प्रारम्भन (Initiation): जीन में प्रमोटर क्षेत्र (promoter) ( अनुलेखन के लिए शुरुआती बिंदु) 5′ सिरे की ओर पाया जाता है प्रमोटर क्षेत्र में TATAAT क्षार क्रम पाये जाते है इसे TATA BOX भी कहते है। इससे RNA POLYMERASE जुडकर DNA के रज्जुक को खोलने का कार्य करता है।
DNA का 3-‘5’ रज्जुक (strain) Template कि तरह काम करता है एवं 5-‘3’ दिशा में RNA का सश्लेषण होता हैं।
दीर्घीकरण(Elongation): आरएनए पोलीमरेज़ DNA के प्रतिअर्थ रज्जुक(Non coding/ template रज्जुक) का उपयोग करके NTP (nucleoside triphosphate) जोडकर एक पूरक आरएनए रज्जुक को संश्लेषण करता है।
समापन (Termination): आरएनए पोलीमरेज़ जब जीन के समापन क्षेत्र (terminator)तक पहुंचता है तो समापन कारक Rho factor DNA के रज्जु से जुडता है तथा DNA, RNA तथा RNA Polymerase को पृथक कर देता है।
DNA प्रतिकृति
डीएनए के द्वारा अपने समान नया डीएनए बनाने की प्रक्रिया को डीएनए प्रतिकृति कहते है।
DNA की प्रतिकृति चरणों में पूरी होती है –
पराकुण्डलन को खोलना (Uncoiling of Supercoiling):-
DNA कुंडली में पाये जाने वाले पराकुण्डलन (Spuercoiling) को Topoisomerase द्वारा अकुंडलित किया जाता है। Topoisomerase Enzymeमें डीएनए रज्जुक (Strand) काटने (Nuclease) तथा उनको पुनः जोड़ने (Ligase) की क्षमता होती है। Topoisomerase दो प्रकार का होता है। –

Topoisomerase-I
Topoisomerase-II – E.coli में इसे DNA gyrase भी कहते है।
2. फॉस्फॉलिरिकरण (Phosphorylation):-
डीएनए के संश्लेषण (synthesis) में काम आने वाले न्यूक्लिओटाइड में केवल एक ही फॉस्फेट समूह जुडा होने के कारण मोनोफॉस्फेट के रूप में होता है। डीएनए के सभी चार प्रकार के मोनोफॉस्फेट ATP से 2-2 अणु फॉस्फेट के लेकर ट्राई फॉस्फेट बनाते है। इसे फॉस्फॉलिरिकरण (Phosphorylation) कहते है। यह प्रक्रिया फोस्फोराइलेज (Phosphorilase) एंजाइम की उपस्थिति में होती है। इससे चार प्रकार के डिऑक्सीन्यूक्लिओटाइड ट्राईफॉस्फेट (dNTP) बनते है

- डिऑक्सीएडिनोसिन ट्राईफॉस्फेट (dATP)
- डिऑक्सीग्वानोसिन ट्राईफॉस्फेट (dGTP)
- डिऑक्सीसाईटीडीन ट्राईफॉस्फेट (dCTP)
- डिऑक्सीथायमिडीन ट्राईफॉस्फेट (dTTP)
3. आरंभन स्थल (Replication Origin Site):-
DNA प्रतिकृति की क्रिया डीएनए के कुछ विशिष्ट स्थानों से शुरू होती जिनको आरंभन स्थल, समारम्भन स्थल या द्विगुणन मूल कहते है। एक ही शब्द का हिंदी में अलग-अलग अनुवाद किया जाता है इसलिए में आपको English शब्दों पर भी ध्यान देने का सुझाव दुगा। प्रोकेरियोट कोशिका में केवल एक ही आरंभन स्थल (Replication Origin Site) होता है। जो कोशिका झिल्ली से जुड़ा रहता है। इसे Ori-C कहते है। जबकि युकेरियोट में बहुत सारे आरंभन स्थल होते है। जिसके कारण युकेरियोट में प्रतिकृति कई स्थानों से शुरू होती है।
4. डीएनए कुण्डली का पृथक्करण (Unwinding of DNA double helix):-
डीएनए के दोनों रज्जुक (Strand) को पृथक करने का कार्य Helicase Enzyme द्वारा किया जाता है। रज्जुक को लड़ी, वलयक आदि भी कहा जाता है।
Helicase आरंभन स्थल से जुड़कर दोनों रज्जुको के क्षार युग्म के Hydrogen बंध को तोड़कर दोनों रज्जुक (Strand) को अलग कर देता है। इस एंजाइम को Origin Binding Protein भी कहा जाता है।
जब Helicase दोनों रज्जुको को अलग करने के बाद दोनों रज्जुको को स्थिरता प्रदान करने तथा फिर से कुंडलित होने से रोकने के लिए DNA Binding Protein (DBP) अलग हुए दोनों रज्जुक (Strand) से जुड़ जाती है। इस प्रोटीन को Single Stranded Binding protein (SSB Protein) या हेलिक्स स्थाई कारक प्रोटीन (Helix Stabilising Protein) भी कहते है।
डीएनए के दोनों पृथक रज्जुक (Strand) Y आकार की संरचना बनाते है। जिसे प्रतिकृति द्विशाखा (Replication Fork) कहते है।
5. नये डीएनए रज्जुक का संश्लेषण (Synthesis of New DNA Strand):-
डीएनए रज्जुक (DNA Strand) पर नये रज्जुक (Strand) का निर्माण डीएनए पालीमरेज एंजाइम द्वारा किया जाता है। लेकिन यह बहुलकीकरण (Polymerization) की शुरुआत नहीं कर सकता इसलिए DNA निर्भर RNA Polymerase (DNA dependent RNA Polymerase) के द्वारा 60-70 न्यूक्लिओटाइड युक्त छोटे पूरक लड़ी (Complementary Chain) का निर्माण किया जाता है। जिसे RNA Primer कहते है। जिससे बहुलकीकरण (Polymerization) की शुरुआत होती है।
RNA Primer बनने के बाद DNA Polymerase एंजाइम इसके 3’ सिरे के हाइड्रोक्सिल (-OH) से नये dNTP को जोड़कर 5’-3’ की दिशा में नये डीएनए का संश्लेषण (synthesis) करते है। याद रखे की DNA Polymerase एंजाइम केवल 5’-3’ दिशा में ही बहुलकीकरण (Polymerization) करता है।
6. सतत एवं असतत DNA संश्लेषण (Continuous and Discontinuous DNA Synthesis)
DNA Polymerase एंजाइम केवल 5’-3’ दिशा में ही बहुलकीकरण (Polymerization) करता है। इसलिए 3’-5’ रज्जुक (Strand) पर नए सतत रज्जुक (Continuous Strand) का संश्लेषण (synthesis) 5’-3’ दिशा में होता है। जबकि 5’-3’ रज्जुक (Strand) पर छोटे –छोटे RNA Primer का निर्माण होता है। जिससे 3’-5’ दिशा में असतत रज्जुक (Discontinuous Strand) का संश्लेषण (synthesis) होता है। इस असतत डीएनए रज्जुक (Discontinuous Strand) को ओकाजाकी खण्ड (Okazaki Fragment) कहते है। ये ओकाजाकी खण्ड सिर्फ RNA Primer के बने होते है।
7. अग्रगामी एवं पश्चगामी रज्जुक (Leading and Lagging Strand):-
DNA के सतत रज्जुक (Strand) को अग्रगामी रज्जुक (Leading Strand) कहते। इसमें केवल एक ही RNA Primer होता। इनका निर्माण Replication Fork की तरफ होता है। तथा असतत डीएनए रज्जुक (Discontinuous DNA Strand) को पश्चगामी रज्जुक (Lagging Strand) कहते है। इसमें कई RNA primers होते है। इनका निर्माण Replication Fork से विपरीत दिशा में होता है। RNA primers को DNA Polymerase-I द्वारा पृथक किया जाता है। और DNA ligase द्वारा इन खण्ड को जोड़ दिया जाता है।
8. DNA प्रतिकृति की समाप्ति (Termination of DNA Replication):-
E.coli में DNA प्रतिकृति प्रक्रिया की समाप्ति में लिए समापक अनुक्रम (Terminating Sequence) पाये जाते है। जिनको Ter site कहते है। DNA प्रतिकृति की समाप्ति के लिए Tus Protein सहायता करती है।
प्रोकेरियोट तथा युकेरियोट दोनों में DNA प्रतिकृति की द्विदिशात्मक (Bidirectional) होता है।
DNA Polymerase के प्रकार –
प्रोकेरियोट में DNA Polymerase तीन प्रकार का होता है –
(1) DNA Polymerase-I
इसे DNA Polymerase-A भी कहा जाता है। ये ओकाझाकी खंडो को हटा कर नये डीएनए के संश्लेषण (synthesis) का काम करता है। ये Proofreading (सम्भावित संशोधन हेतु DNA अनुक्रम को पढ़ना) का कार्य करता है। इसे कोर्नबर्ग एंजाइम (Kornberg Enzyme) भी कहते है।

(2) DNA Polymerase-II
इसे DNA Polymerase-B भी कहा जाता है। ये डीएनए की मरम्मत का कार्य करता है।

(3) DNA Polymerase-III
इसे DNA Polymerase-C भी कहा जाता है। ये डीएनए रज्जुक (Strand) का दीर्घीकरण (Elongation) करता है। यह भी Proofreading का कार्य करता है।

युकेरियोट में DNA Polymerase पांच प्रकार का होता है। –
(1) DNA Polymerase-α
ये प्रतिकृति की शुरुआत करता है। इसमें Primase क्षमता होती है।
(2) DNA Polymerase-β
ये डीएनए की मरम्मत का कार्य करता है।
(3) DNA Polymerase-γ
ये mt-DNA की प्रतिकृति बनता है।
(4) DNA Polymerase-δ
ये डीएनए संश्लेषण (synthesis) में भाग लेता है। और डीएनए रज्जुक (Strand) का दीर्घीकरण (Elongation) करता है।
(5) DNA Polymerase-ε
ये डीएनए की मरम्मत का कार्य करता है।
SSB- Replication Protein A also called as Rp-A
PCNA- Proliferating Cell Nuclear Antigen
Rf-C – Replication factor- C
आरएनए की संरचना, प्रकार तथा उनके कार्य
RNA का पूरा नाम राईबोज न्यूक्लिक अम्ल होता (Ribose Nucleic Acid ) है। यह एकल पॉलीराईबोन्यूक्लियोटाइड श्रृंखला (Single Polyribonucleotide Chain) का बना होता है। इसमें राईबोन्यूक्लियोसाइड (Ribonucleoside) तथा राईबोन्यूक्लियोटाइड (Ribonucleotide) होते है। राईबोन्यूक्लियोटाइड चार प्रकार के होते है –
- एडीनिलिक अम्ल Adenylic Acid (AMP)
- ग्वानिलिक अम्ल Guanylic Acid (GMP)
- साईटीडीलिक अम्ल Cytidylic Acid (CMP)
- युरिडीलिक अम्ल Uridylic Acid (UMP)
RNA तीन प्रकार का होता है –

1. mRNA या संदेशवाहक RNA (Messenger RNA): –
mRNA या संदेशवाहक RNA प्रोटीन संश्लेषण के लिये केन्द्रक (Nucleus) में स्थित DNA से प्रोटीन संश्लेषण के स्थान तक सूचना प्राप्त करता हैं। mRNA का निर्माण DNA के रज्जु को टेम्पलेट के रूप में प्रयोग करके किया जाता है। जिसके कारण mRNA में पूरक क्षार (Complimentory Base) होते है। एक कोशिका में कुल RNA का 3-5% भाग t-RNA होता है।

प्रोकेरियोटिक कोशिकाओं में एक ही mRNA से एक से अधिक पॉलीपेप्टाइड श्रृंखलाओं (Polypeptide Chain) का निर्माण होता है। इसलिए इन mRNA को पॉलीसिस्ट्रोनिक (Polycistronic) RNA कहते है। जबकि युकेरियोटिक कोशिका में एक mRNA से केवल एक ही पॉलीपेप्टाइड श्रृंखला बनाने के संश्लेषण के लिए सूचनाएँ होती है। जिसे मोनोसिस्ट्रोनिक (Monocistronic) RNA कहते है।
2. tRNA या ट्रांसफर RNA (Transfer RNA) :-
यह सबसे छोटा RNA है। जिसमें 75-95 राईबोन्यूक्लियोटाइड पाये जाते है। यह प्रोटीन के संश्लेषण के समय राइबोसोम के A-स्थल में अमीनो अम्ल का स्थानांतरण करता है। प्रत्येक 20 amino acids के लिए विशिष्ट tRNA होते है। प्रत्येक tRNA एक ही अमीनो अम्ल के लिये विशिष्ट होता है। और प्रोटीन संश्लेषण के लिए अमीनो अम्ल को कोशिका द्रव्य से राइबोसोम में ले जाता है। अतः एक कोशिका में tRNA या ट्रांसफर RNA को घुलनशील (Soluble RNA) या Adaptor RNA भी कहा जाता है। एक कोशिका में कुल RNA का 16-18% भाग t-RNA होता है।

रोबर्ट हॉली ने tRNA की क्लोवर लीफ (Clover Leaf) संरचना प्रस्तुत की जिस के अनुसार tRNA में चार भुजाएँ होती है –
ग्राही भुजा (Acceptor Arm) –
इस भुजा से 5’ तथा 3’ सिरे होते है। 3’ सिरे पर CCA (साइटोसिन-साइटोसिन एडेनिन) क्षार होते हैं तथा 5’ सिरे पर G(ग्वानिन) होता है।। अमीनो अम्ल का कार्बोक्सिल समूह (-COOH) है। 3’ सिरे पर CCA के एडेनोसिन के द्वारा जुड़ता है।
प्रतिकोडॉन भुजा (Anticodon Arm) –
यह भुजा ग्राही भुजा के विपरीत भुजा होती है। इस पर तीन क्षार का एक विशेष अनुक्रम होता है। जिसे प्रतिकोडॉन कहते है। ये प्रतिकोडॉन mRNA पर स्थित कोडॉन के पुरक होते है। जैसे यदि mRNA पर AUG कोडॉन होते है तो tRNA UAG कोडॉन होंगे।
TψC भुजा (TψC Arm) –
यह भुजा tRNA को राइबोसोम से जोड़ने में सहायता करती है।
DHU भुजा (DHU Arm) –
यह भुजा एंजाइम से जुड़ने में सहायता करती है। यह DHU का पूरा नाम डाइहाइड्रोयूरिडीन है, जो एक असामान्य क्षार है।
tRNA में कुछ असामान्य क्षार होते हैं जैसे इनोसिन (Inosine) व डाइहाइड्रोयूरिडीन (Dihydrouridine) आदि।
3. rRNA या राइबोसोमल RNA (Ribosomal RNA): –

- rRNA राइबोसोम का एक घटक है। यह Ribosome का 80% भाग बनता है। राइबोसोम राइबोन्यूक्लियो प्रोटीन तथा rRNA से मिलकर बना होता है।। rRNA का संश्लेषण क्रोमोसोम के न्यूक्लिओलर आर्गनाइजर क्षेत्र में पायी जाने वाली राइबोसोमल जीनों में निहित सूचना से होता है।। एक कोशिका में पाये जाने वाले कुल RNA का 80% भाग rRNA होता है।
- प्रोकेरियोटिक कोशिका, माइटोकांड्रिया, तथा लवक में 70s प्रकार का ribosome पाया जाता है। जो दो उप-इकाइयों 50s तथा 30s से मिलकर बना होता है।
- 50s राइबोसोम में 23s rRNA, 5s rRNA होता है। जबकि 30s राइबोसोम में 16s rRNA होता है।
- 23s rRNA में लगभग 3000 राईबोन्यूक्लियोटाइड, 5s rRNA में लगभग 120 राईबोन्यूक्लियोटाइड 16s rRNA 1500 राईबोन्यूक्लियोटाइड पाये जाते है।
- युकेरियोटिक कोशिका में 80s प्रकार का ribosome पाया जाता है। जो दो उप-इकाइयों 60s तथा 40s से मिलकर बना होता है।
- 60s राइबोसोम में 28s, 5.8s तथा 5s rRNA होता है। जबकि 40s राइबोसोम 18s rRNA होता है। 28s rRNA में 5000 राईबोन्यूक्लियोटाइड, 5.8s rRNA में 160 राईबोन्यूक्लियोटाइड, 5s rRNA में 120 राईबोन्यूक्लियोटाइड पाये जाते है। जबकि 18s rRNA में 1800 राईबोन्यूक्लियोटाइड होते है।
- rRNA संरचनात्मक अणु (Structural Molecule) है। जबकि tRNA तथा rRNA क्रियात्मक अणु (Functional Molecule) है।
- राइबोसोम की इकाइयों व इनमें पाये जाने वाले rRNA को स्वेदवर्ग इकाई (Swedberg Unit) (S) द्वारा प्रदर्शित किया जाता है। यह इन अणुओं की अवसादन दरों (Sedimentation Rate) को प्रदर्शित करते है।
अन्य प्रकार के RNA –
- snRNA – Small Nuclear RNA
- scRNA – Small Cytoplasmic RNA
- Ribozyme – RNA एंजाइम की भांति कार्य करता है।
- miRNA – MicroRNA
- piRNA – Piwi-interacting RNA
- snoRNA – Small Nucleolar RNA
- hnRNA – Hetero Nuclear RNA
अनुलेखन (Transcription)
प्रोटीन के निर्माण के लिए डीएनए पर विशिष्ट अनुक्रम (Specific Sequence) पाए जाते हैं। प्रोटीन के निर्माण के लिए आरएनए की आवश्यकता होती है।
अतः DNA को टेम्पलेट (Templet) के रूप में उपयोग करके आरएनए का निर्माण किया जाता है, इस प्रक्रिया को अनुलेखन (Transcription) कहते हैं।
DNA से आनुवंशिक सुचनाओ का RNA में स्थानान्तरण अनुलेखन (Transcription) कहलाता है।
आइए अनुलेखन (Transcription) के बारे में ओर अधिक जानकारी प्राप्त करते हैं। अ
नुलेखन (Transcription) की प्रक्रिया कोशिका के केंद्रक में होती है। यह प्रक्रिया कोशिका चक्र के G1 तथा G2 अवस्था में होती हैं।
अनुलेखन इकाई (Transcriptional Unit):-
DNA का वह खंड (Fragment) जिसके अनुलेखन (Transcription) से केवल एक आरएनए का निर्माण होता है। अनुलेखन इकाई (Transcriptional Unit) कहलाता हैं।
अनुलेखन इकाई (Transcriptional Unit) के तीन भाग होते हैं –
- प्रारंभन स्थल
- समापक स्थल
- संरचनात्मक जीन
प्रारंभन स्थल (Initation Site):-
DNA का वह क्षेत्र जहां से अनुलेखन (Transcription) की प्रक्रिया प्रारंभ होती है उसे प्रारंभन स्थल कहते हैं।
प्रारंभन स्थल के पहले प्रमोटर स्थल (Promotor) होता है। प्रमोटर स्थल को उन्नायक भी कहा जाता है। इस स्थल में 20 से 200 न्यूक्लियोटाइड पाए जाते हैं।
सजीवों में दो प्रकार के प्रमोटर स्थल होते है-
TTGACA:-
प्रथम TTGACA स्थल होता है। इस स्थल से आरएनए पोलीमरेज़ एंजाइम का सिग्मा कारक ( σ Factor) जुड़ता है। इसमें 35 न्यूक्लियोटाइड पाए जाते हैं। इसको पहचान स्थल (Recognition Sequence) भी कह सकते है।
Pribnow Box:-
दूसरा Pribnow बॉक्स होता है। जिसमें TATAAT अनुक्रम होते हैं। इसी स्थान से आरएनए पोलीमरेज़ एंजाइम जुड़ता है। इसमें 10 न्यूक्लियोटाइड पाए जाते हैं। इसे TATA BOX तथा Hogness Box भी कहते है।
समापक स्थल (Terminator Site):-
समापक वह स्थल होता है, जहां अनुलेखन (Transcription) की प्रक्रिया समाप्त हो जाती है। यह टेम्पलेट रज्जुक के 3’ सिरे की ओर होता है।
संरचनात्मक जीन (Structural Gene):-
प्रारंभिक स्थल तथा समापक स्थल के बीच संरचनात्मक जीन होती है। इसमें RNA coding sequence होते है। इस संरचनात्मक जीन के 5’- 3’ रज्जुक को नॉन-टेम्पलेट (Templet) रज्जुक रज्जुक तथा 3’- 5’ रज्जुक को टेम्पलेट (Templet) रज्जुक कहते हैं।
टेम्पलेट (Templet) रज्जुक के अनुक्रम का उपयोग करके उसके पूरक अनुक्रम (Complementary Sequence) वाला आरएनए बनता है। टेम्पलेट (Templet) रज्जूक को नॉन कोडिंग रज्जुक भी कहते हैं, तथा नॉन-टेम्पलेट (Templet) रज्जुक को कोडिंग रज्जुक भी कहते है।
अनुलेखन की क्रियाविधी (Mechanism of Transcription):–
प्रारम्भन (Initiation)
- जीन में प्रमोटर स्थल (promoter) ( अनुलेखन के लिए शुरुआती बिंदु) टेम्पलेट रज्जुक के 5’ सिरे की ओर पाया जाता है।
- DNA के प्रमोटर स्थल की पहचान करके सिग्मा कारक ( σ Factor) उस से जुड़ जाता है और आरएनए पोलीमरेज़ एंजाइम DNA से जुड़कर उसकी कुंडलियों के नाइट्रोजन क्षार के मध्य पाए जाने वाले हाइड्रोजन बंध को तोड़ने का कार्य करता है।
- DNA का 3’-‘5’’ रज्जुक (strain) Template कि तरह काम करता है एवं 5’-‘3’’ दिशा में RNA का सश्लेषण होता हैं।
दीर्घीकरण(Elongation):
जब दोनों रज्जुक (strain) अलग हो जाते हैं। तो आरएनए पोलीमरेज़ DNA के प्रतिअर्थ रज्जुक(Non coding/ template रज्जुक) का उपयोग करके NTP (nucleoside triphosphate) ( इनको राइबोन्यूक्लियोटाइड ट्राई फास्फेट भी कहते है) जोडकर एक पूरक आरएनए रज्जुक (strain) का संश्लेषण करता है।
राइबोन्यूक्लियोटाइड ट्राई फॉस्फेट निम्न प्रकार के होते है –
- यूरिडीनट्राईफॉस्फेट (UTP)
- ग्वानोसिनट्राईफॉस्फेट (GTP)
- साइटोसिनट्राईफॉस्फेट (CTP)
- एडिनिनट्राईफॉस्फेट (ATP)
आरएनए पोलीमरेज़ राइबोन्यूक्लियोटाइड ट्राई फॉस्फेट का उपयोग करके उनको मोनो फॉस्फेट के रूप में जोड़ता है।
आरएनए पोलीमरेज़ DNA पोलीमरेज़ की तरह भी केवल 5’-3’ दिशा में कार्य करता है अतः यह 5’-3’ दिशा में ही RNA का संश्लेषण कर सकता है।
केवल टेम्पलेट (Templet) रज्जुक जो कि 3’-5’ दिशा में होता है पर ही नए आरएनए का संश्लेषण होता है।
समापन (Termination):
आरएनए पोलीमरेज़ जब जीन के समापन क्षेत्र (terminator)तक पहुंचता है। तो समापन कारक Rho factor DNA के रज्जुक से जुडता है तथा DNA, RNA व RNA Polymerase को पृथक कर देता है। इस प्रकार एक नए आरएनए का निर्माण होता है।
E.coli में समापन की प्रक्रिया Rho factor की अनुपस्थिति में होती है जिसे Rho Independent Termination भी कहते है।
आरएनए पोलीमरेज़ की संरचना:-
RNA पोलीमरेज़ में कुल 5’ पॉलीपेप्टाइड इकाइयां होती है। जिनमें दो α, एक β, एक β’ तथा एक सिग्मा कारक ( σ Factor) होता है।
RNA पोलीमरेज़ तीन प्रकार का होता है-
आरएनए पोलीमरेज़-I
यह केन्द्रिक में पाया जाता है, इसके द्वारा rRNA का निर्माण होता है
आरएनएपोलीमरेज़- II
यह केंद्रक में पाया जाता है इसके द्वारा mRNA का निर्माण होता है
आरएनए पोलीमरेज़- III
यह tRNA का निर्माण करता है
इसको आप RMT से याद कर सकते हो जहां यानी R- rRNA, M-mRNA, T-tRNA जो एक क्रम में है।
पश्च अनुलेखन रूपान्तरण (Post-Transcription Modification)
Prokaryote में,अनुलेखन (Transcription) और अनुवादन (Translation) की प्रकिया दोनों एक ही समय में पूरी होती हैं। अनुलेखन (Transcription) होने से पहले ही अनुवादन (Translation) प्रारंभ हो जाता है। यूकेरियोट्स में, अनुवादन (Translation) प्रारंभ होने से पहले प्राथमिक आरएनए / पूर्व RNA (Pre-RNA) को संशोधित किया जाता है। इस चरण को RNA प्रसंस्करण (RNA processing) या अनुलेखन (Transcription) संशोधन या पश्च अनुलेखन रूपान्तरण (Post-Transcription Modification) कहा जाता है। RNA को केन्द्रक छिद्रों के माध्यम से साइटोप्लाज्म में आना होता है इसलिए RNA को RNAase से सुरक्षा के लिए संशोधित किया जाता है। सभी यूकेरियोटिक mRNA तथा बैक्टीरिया और यूकेरियोट दोनों के tRNA का प्रसंस्करण (Processing) होता है।
एक यूकेरियोटिक जीन में कोडिंग क्षेत्रों के साथ-साथ कई गैर-कोडिंग क्षेत्र भी पाये जाते है। कोडिंग क्षेत्रों को एक्सॉन या व्यक्तेक (Exon) तथा गैर-कोडिंग क्षेत्र को इंट्रॉन या अव्यक्तेक (Intron) कहते है। एक्सॉन के द्वारा प्रोटीन का कूटलेखन (Code) होता है, जबकि इंट्रॉन के द्वारा प्रोटीन का कूटलेखन(Code) नहीं होता। जब एक mRNA निर्माण होता है, तो उसमे इंट्रॉन तथा एक्सॉन दोनों ही पाए जाते है जिसे hnRNA (Heteronuclear RNA) कहते है। इस hnRNA का संशोधन के पश्चात एक mature mRNA का निर्माण होता है RNA संशोधन की प्रकिया निम्न चरणों में होती है-
(1) 5’ सिरे पर आच्छादन (Addition of a Cap on the 5’ end)
(2) 3’ सिरे पर पुच्छन (Addition of a Poly-A tail on the 3’ end)
(3) संबंधन (Splicing)
आच्छादन (5’ Cappping) –
mRNA अत्यंत अस्थायी होता है। राइबोन्यूक्लिऐज एंजाइम (RNAase) से सुरक्षा के लिए इसके सिरों को सुरक्षित करना आवश्यक होता है इसके लिए guanyl transferase एंजाइम का उपयोग किया जाता है। अनुलेखन (Transcription) या ट्रांसक्रिप्शन (Transcroption) शुरू होने के बाद जैसे ही 20-30 न्यूक्लियोटाइड्स का गठन होता आच्छादन (5’ Cappping) की प्रक्रिया शुरू हो जाती है। आच्छादन (5’ Cappping) प्रक्रिया में,संशोधित ग्वानिन guanine triphosphate (GTP जिसमे एक मिथाइल समूह जुड़ा रहता 7-methyl G) को mRNA के 5’ सिरे पर जोड़ा जाता है।
पुच्छन (3’ Poly A tail):-
यूकेरियोटिक mRNA में 80 से लेकर 250 एडीनोसिन युक्त श्रृंखला को 3’ सिरे पर जोड़ा जाता है जिसे पाली (A) पूंछ कहते है यहाँ A का अर्थ एडीनोसिन है mRNA की पुच्छन (3’ Poly A tail) प्रक्रिया निम्न घटनाओं में सहायता होती है –
1) यह केन्द्रक से परिपक्व mRNA को निर्यात (Export) करने में सहायता करता है।
2) यह mRNA की स्थायित्व (Stability) को बढ़ाता है।
3) यह अनुवादन (Translation) (Translation) के प्रारम्भ के समय अनुवादन (Translation)कारी कारकों (translational factors) के बंधन के लिए पहचान संकेत (recognition signa) के रूप में कार्य करता है।
संबंधन (Splicing):-
इंट्रॉन नॉनकोडिंग अनुक्रम (noncoding nucleotide sequence) है जो जीन में पायी जाती लेकिन प्रोटीन का कूटलेखन (Code) नहीँ करती इंट्रॉन चार प्रकार समूह के होते हैं:
समूह I (Self-splicing Introns): –
इनका संबंधन (Splicing) स्वतः होता है। इनके संबंधन (Splicing) के लिए एंजाइम की आवश्यकता नहीं होती।
समूह II (Self-splicing Introns): –
इनका संबंधन (Splicing) भी स्वतः होता है संबंधन (Splicing) के लिए एंजाइम की आवश्यकता नहीं होती।
स्पलाईसीओसॉमल इंट्रॉन्स (Spliceosomal introns): –
इनका संबंधन (Splicing) स्वतः नहीं होता हैं। संबंधन (Splicing) के लिए एंजाइम की आवश्यकता होती।
इंट्रॉन
जिनके संबंधन (Splicing) के लिए एटीपी की आवश्यकता होती है।
आनुवंशिक कोड

इसकी खोज Nirenberg, Mathai, H.G. Khurana तथा Robert Hali ने की तथा Genetic code शब्द George Gamow ने दिया।
RNA में उपस्थित आनुवंशिक सूचनाओं (Genetic Information) का उपयोग करके एक प्रोटीन का संश्लेषण (Synthesis) करना किसी एक भाषा को दूसरे भाषा में अनुवाद (Translate) करने की तरह है।
प्रोटीन संश्लेषण के दौरान चार नाइट्रोजन क्षार (Nitrogen Base) का उपयोग करके 20 प्रकार के एमिनो अम्लो के लिए कोड़ तैयार किये जाते है।
आनुवंशिक कोड (Genetic Code) डीएनए और बाद में ट्रांसक्रिप्शन द्वारा बने mRNA पर नाइट्रोजन क्षार का विशिष्ट अनुक्रम (Sequence) है, जिनका अनुवाद प्रोटीन के संश्लेषण के लिए एमीनो अम्ल के रूप में किया जाता है।
एक एमिनो अम्ल निर्दिष्ट करने वाले तीन नाइट्रोजन क्षारों के समूह को कोडन, कोड या प्रकुट (Codon) कहा जाता है।
आनुवंशिक कोड की विशेषताए (Specificity of Genetic Code):-
त्रिक प्रकुट या ट्रिपलेट कोड (Triplet Codon):-
mRNA में चार न्यूक्लियोटाइड अनुक्रम (Sequence) (U, G, A तथा C) का इस्तेमाल तीन नाइट्रोजन क्षार युक्त कोडोन (43 = 64) रूप में किया जाता है। अतः कोडोन त्रिक होते है क्योंकि यदि कोडोन एकल होते तो केवल चार ही कोड बनते (41=4) जो 20 प्रकार के एमिनो अम्ल का कूटलेखन नहीं कर सकते और यदि ये द्विक होते तो कुल 16 कोड़ बनते (44=16) जो एमिनो अम्ल को कूटलेखित नहीं कर सकते।

64 कोडो में अर्थ कोडोन्स (Sense Codons) और गैर अर्थ कोडोन्स (Non-sense Codons) शामिल हैं। अर्थ कोडोन्स वे कोडोन होते है, जो एमिनो अम्ल निर्दिष्ट करते हैं। तथा गैर अर्थ कोडोन्स वे है जो एमिनो अम्ल निर्दिष्ट नहीं करते हैं।
अर्थ कोडोन्स की संख्या 61 है। जबकि गैर अर्थ कोडोन्स की संख्या 3 होती है। एक कोडोन के द्वारा केवल एक ही एमिनो अम्ल को कोड़ किया जाता है। लेकिन एक ही एमिनो अम्ल के लिए एक से अधिक कोड होते हैं।
अर्द्धविराम विहीन कोड(Commaless Code): –
आनुवंशिक कोड (Genetic Code) के में बीच कोई विराम चिह्न (Comma or full stop) नहीं होता है। और न ही उनके मध्य कोई रिक्त स्थान (Gap) होता है। आनुवंशिक कोड (Genetic Code) लगातार होते है।

अन-आच्छादित या गैर-ओवरलैपिंग कोड (Non-overlap Code): –
आनुवंशिक कोड (Genetic Code) क्रमिक रूप से तीन के समूह में पढ़ा जाता है। ये एक दुसरे को ओवरलैपिंग नहीं करते है।
एक त्रिक (triplet) का कोई भी नाइट्रोजन क्षार दुसरे त्रिक (triplet) के साथ नहीं पढ़ा जा सकता।
प्रत्येक त्रिक को 5 ‘→ 3’ दिशा से पढ़ा जाता है, इसलिए पहला नाइट्रोजन क्षार 5 ‘ क्षार होता है, उसके बाद मध्यम क्षार होता है उसके के बाद अंतिम क्षार होता है जो 3’ नाइट्रोजन क्षार है।

अनुवादन (Translation)
अनुवादन वह प्रक्रिया है। जिसमें mRNA पर उपस्थित जेनेटिक कोड का उपयोग करके प्रोटीन का निर्माण किया जाता है। अनुवादन की प्रक्रिया अंतः प्रद्रव्यी जलिका (Endoplasmic Reticulum) पर उपस्थित राइबोसोम में संपन्न होती है। अनुवादन की प्रक्रिया में जीवद्रव्य में उपस्थित अमीनो अम्ल का उपयोग करके राइबोसोम द्वारा पॉलीपेप्टाइड श्रंखला (Polypeptide Chain) का निर्माण किया जाता है।
इसकी प्रक्रिया निम्न चरणों में संपन्न होती है-
अमीनो अम्ल का सक्रियण (Activation of Amino Acids)
जीवद्रव्य में एमिनो एसाइल सिंथेटेज (Aminoacyl Synthetase) एंजाइम की उपस्थिति में एक अमीनो अम्ल ATP से जुड़ता है। इस अभिक्रिया में Mg2+ सहायता करता है। इस अभिक्रिया के फलस्वरुप एमिनो एसाइल एडीनाइलेट सम्मिश्र (Aminoacyl Adynylate Complex)का निर्माण होता है।

Amino Acid + ATP Aminoacyl Adynylate Complex
अमीनो अम्ल का tRNA पर स्थानांतरण (Transfer of Amino Acids on tRNA)
एमिनो एसाइल एडीनाइलेट सम्मिश्र (Aminoacyl Adynylate Complex) एक विशिष्ट प्रकार के tRNA के साथ जुड़ती है। जिससे आवेशित tRNA (Charged tRNA) का निर्माण होता है। और AMP का निष्कासन होता है। अमीनो अम्ल tRNA के 3’ सिरे पर उपस्थित CCA अनुक्रम से जुड़ता है।

Aminoacyl Adynylate Complex + tRNA → Activated tRNA + AMP + Aminoacyl synthetase
अनुवादन का आरंभन (Initiation of Translation)
अनुवादन के आरंभन के लिए राइबोसोम की छोटी इकाई mRNA के साथ जुड़ती है तथा mRNA पर उपस्थित प्रारंभिक कोडोन AUG की पहचान करके methionine अमीनो अम्ल युक्त tRNA प्रारंभिक कोडोन से हाइड्रोजन बंध के द्वारा जुड़ जाता है। जिसमें GTP के द्वारा ऊर्जा प्रदान की जाती है। अब राइबोसोम की छोटी इकाई और mRNA के साथ राइबोसोम की बड़ी इकाई भी जुड़ जाती है। इस दौरान AUG P-स्थल में होता है।
अनुवादन का दीर्घीकरण (Elongation of Translation)
एक नया आवेशित tRNA (Charged tRNA) GTP की उपस्थिति में राइबोसोम के A- स्थल में उपस्थित कोडोन से जुड़ जाता है। इसके पश्चात P-स्थल में उपस्थित tRNA के अमीनो अम्ल A- स्थल में उपस्थित अमीनो अम्ल के साथ पेप्टाइड बंध (Peptide Bond) बनाता है। जिसको पेप्टीडाइल ट्रांसफेरेज (Peptidyl Transferase) एंजाइम द्वारा उत्प्रेरित किया जाता है।
P- स्थल के tRNA पर उपस्थित अमीनो अम्ल का स्थानांतरण A-स्थल में उपस्थित tRNA पर हो जाता है। जब राइबोसोम mRNA पर 5’-3’ दिशा में गति करता है। तो P-स्थल में उपस्थित अनावेशित (Uncharged) tRNA E-स्थल में आ जाता है। और A-स्थल का tRNA P-स्थल में आ जाता है। इसी प्रकार A-स्थल में नया आवेशित tRNA प्रवेश करता है। और E-स्थल में उपस्थित अनावेशित tRNA राइबोसोम से बाहर निकल जाता है। यह प्रक्रिया लगातार चलती रहती है। इस प्रकार एक नए पॉलीपेप्टाइड श्रृंखला का संश्लेषण होता है।
अनुवादन का समापन (Termination of Translation)
जब राइबोसोम mRNA पर गति करता हुआ समापक (Stop) कोडोन (UAG,UGA,UAA) पर पहुंचता है। तो राइबोसोम के A-स्थल में कोई tRNA प्रवेश नहीं करता। जिसके कारण अनुवादन की प्रक्रिया समाप्त हो जाती है। जैसे ही अनुवादन की प्रक्रिया समाप्त होती है। पॉलीपेप्टाइड श्रृंखला मुक्त हो जाती है और mRNA तथा राइबोसोम पृथक हो जाते है।
अनुवादन में काम आने वाले प्रोटीन कारक
इनको अनुवादन कारक भी कहा जाता है। ये तीन प्रकार के होते है –
- आरंभन कारक (Initiation Factors)
- दीर्घीकरण कारक (Elongation Factors)
- समापन कारक (Termination or Release Factors)
आरंभन कारक (Initiation Factors)
ये तीन प्रकार के होते है –
- IF-1 – ये IF-3 के बंधने में सहायता करता है।
- IF-2 – ये 30S उप-इकाई के साथ GTP तथा tRNA को जोड़ता है।
- IF-3 – ये 30S उप-इकाई से जुड़ता है और समय से पूर्व 50S उप-इकाई को जोड़ने से रोकता है।
दीर्घीकरण कारक (Elongation Factors)
ये तीन प्रकार के होते है –
- EF-Tu – GTP तथा एमिनो एसाइल-tRNA को जोड़ता है।
- EF-Ts – EF-Tu से GTP को अलग करता है।
- EF-G – Translocation को बढ़ावा देता है।
समापन कारक (Termination or Release Factors)
ये चार प्रकार के होते है –
- RF-1 – UAA और UAG की पहचान करता है।
- RF-2 – UAA और UGA की पहचान करता है।
- RF-3 – RF-1 तथा RF-2 की सहायता करता है।
- RRF – EF-G के साथ मिलकर राइबोसोम की दोनों इकाईयों को अलग करता
अनुवादन की प्रक्रिया को रोकने वाले कारक –
निम्न एंटीबायोटिक द्वारा अनुवादन की प्रक्रिया को रोका जाता है-
- Chloramphenicol
- Streptomycin
- Puromycin
- Tertacycline
- Paromomycin
- Fusidic Acids
- Erythromycin
- Cycloheximide
- Diphtheria toxin
Class 12 Biology All Chapter Notes and Que & Ans
- Class 12 Biology Chapter 1 जनन (Reproduction) Notes in Hindi
- Class 12 Biology Chapter 1 जनन (Reproduction) Question Answer in Hindi
- Class 12 Biology Chapter 2 पुष्पीय पादपो में लैंगिक जनन Notes in Hindi
- Class 12 Biology Chapter 2 पुष्पीय पादपो में लैंगिक जनन Question Answer in Hindi
- Class 12 Biology Chapter 3 शुक्राणु की संरचना एवं प्रकार तथा वीर्य Notes in Hindi
- Class 12 Biology Chapter 3 शुक्राणु की संरचना एवं प्रकार तथा वीर्य Question Answer in Hindi
- Class 12 Biology Chapter 4 जनन स्वास्थ्य (Reproductive Health) Notes in Hindi
- Class 12 Biology Chapter 4 जनन स्वास्थ्य (Reproductive Health) Question Answer in Hindi
- Class 12 Biology Chapter 5 वंशागति तथा विविधता के सिद्धांत Notes in Hindi
- Class 12 Biology Chapter 5 वंशागति तथा विविधता के सिद्धांत Question Answer in Hindi
- Class 12 Biology Chapter 6 वंशागति का आणविक आधार in Hindi Notes
- Class 12 Biology Chapter 6 वंशागति का आणविक आधार in Hindi Question Answer
- Class 12 Biology Chapter 7 विकास in Hindi Notes
- Class 12 Biology Chapter 7 विकास in Hindi Question Answer
- Class 12 Biology Chapter 8 प्रतिरक्षा तंत्र की कोशिकाएं Notes in Hindi
- Class 12 Biology Chapter 8 प्रतिरक्षा तंत्र की कोशिकाएं Question Answer in Hindi
- Class 12 Biology Chapter 9 खाद्य उत्पादन में वृद्धि की कार्यनीति Notes in Hindi
- Class 12 Biology Chapter 9 खाद्य उत्पादन में वृद्धि की कार्यनीति Question Answer in Hindi
- Class 12 Biology Chapter 10 मानव कल्याण में सूक्ष्म जीव Notes in Hindi
- Class 12 Biology Chapter 10 मानव कल्याण में सूक्ष्म जीव Question Answer in Hindi
- Class 12 Biology Chapter 11 जैव प्रौद्योगिकी-सिद्धांत व प्रक्रम Notes in Hindi
- Class 12 Biology Chapter 11 जैव प्रौद्योगिकी-सिद्धांत व प्रक्रम Question Answer in Hindi
- Class 12 Biology Chapter 12 जैव प्रौद्योगिकी एवं उसके उपयोग Notes in Hindi
- Class 12 Biology Chapter 12 जैव प्रौद्योगिकी एवं उसके उपयोग Question Answer in Hindi
- Class 12 Biology Chapter 13 जीव और समष्टियाँ Notes in Hindi
- Class 12 Biology Chapter 13 जीव और समष्टियाँ Question Answer in Hindi
- Class 12 Biology Chapter 14 पारितंत्र Notes in Hindi
- Class 12 Biology Chapter 14 पारितंत्र Question Answer in Hindi
- Class 12 Biology Chapter 15 जीव विविधतता एवं संरक्षण Notes in Hindi
- Class 12 Biology Chapter 15 जीव विविधतता एवं संरक्षण Question Answer in Hindi
- Class 12 Biology Chapter 16 पर्यावरण के मुद्दे Notes in Hindi
- Class 12 Biology Chapter 16 पर्यावरण के मुद्दे Question Answer in Hindi

